基于并行计算优化的WindSTORM PLUS算法  下载: 1034次
下载: 1034次
1 引言
超分辨显微成像技术具有突破光学衍射极限的特性,可对细胞内的超精细结构进行清晰成像,已成为生命科学领域强有力的研究手段之一[1-4]。光学重建显微镜(STORM)[5]作为一种单分子定位显微成像技术,相对于其他超分辨显微成像(如受激发射损耗显微镜成像等[6-7]),具有成像系统结构复杂度低、分辨率高等优点。随着直接随机光学重建显微镜(dSTORM)[8]的出现,荧光分子的闪烁条件被进一步降低,STORM被进一步推广应用。STORM成像效果不仅受限于荧光分子的闪烁条件,也与定位重建和数据处理的方法息息相关[9]。随着科研级互补金属氧化物半导体(sCMOS)等传感器在超分辨显微成像领域的逐渐应用[10-11],STORM也面临着大视场下大数据集高速处理的挑战。
自STORM技术于2006年被提出以来,单分子定位重建的每一个步骤都涌现出了各类预处理及拟合方法,如空间滤波和小波滤波等滤波算法[12-13]、局部最大值和非极大值抑制等分子检测算法[14]、迭代高斯拟合和非迭代的压缩感知等定位点拟合算法[13,15];而且这些算法集成在了不同的单分子定位处理软件中,如FALCON、DAOSTORM和Peak Fit等[16-19]。Ovesny等[20]在2014年发表的开源ThunderSTORM软件中集成了多种滤波及定位算法,使得该软件在STORM领域得到广泛应用。2018年,Martens等[21]提出的Phasor法使单分子单拟合速度达到MHz级别,并将其补充进了ThunderSTORM软件中。2015年和2019年,洛桑联邦理工学院的生物医学成像小组[22-23]对已有的30多款单分子定位软件进行了对比,分析了它们在不同条件下的定位准确性,为超分辨定位的数据处理提供了参考,但此对比集中在小视场数据集下(例如128×128)。2019年,Ma等[24]基于频域卷积滤波及梯度定位的思想,提出了非迭代计算的WindSTORM,其在数据处理速度上相比于传统的迭代算法提高了2~3个数量级,尤其适合用于复杂噪声下的超分辨图像处理;但目前该算法只适用于固定数据尺寸(例如2N)的数据,且对计算机的内存要求较高,例如,内存为256 GB的计算机单次最大数据处理能力约为8 GB。
为了解决上述算法存在的不足,并迎合大视场范围下的超大数据集处理,本文构建了WindSTORM PLUS算法;该算法可对数据集实现智能分组以及计算模式的选择,并因采用并行加速计算而大幅提升了重建速度。本文在高低密度和各尺寸的模拟数据集下进行了验证实验,实验结果表明,在512×512和1024×1024大尺寸成像范围的超大数据集(例如,5~20 GB)下,相比WindSTORM和ThunderSTORM中的最小二乘法高斯拟合(Gauss-WLS),WindSTORM PLUS将定位速度提高了1000%;相比Phasor,WindSTORM PLUS将定位速度提高了50%。此外,本文基于easySTORM[25]方法搭建了STORM成像系统并进行了实验,实验结果表明,WindSTORM PLUS的耗时只有WindSTORM和Gauss-WLS的9%,Phasor拟合方法的70%左右,表明了所提算法在STORM数据处理速度上的优越性。本文构建的WindSTORM PLUS开源算法为单分子定位的超分辨图像提供了一个全新的高速处理方案,特别适合用于大成像视场下采集的超大数据集重组。
2 WindSTORM PLUS算法与模拟验证
2.1 WindSTORM算法
WindSTORM是一套用于二维STORM超分辨数据处理的算法[24],该算法的流程主要分为四部分:1)基于时间极小值和荧光衰减因子进行背景的估计与去除;2)结合频域与光学传递函数的去卷积频率滤波,恢复重叠区域的环绕发射点;3)根据重叠区域环绕发射点确定重叠区域中心发射点并对重叠区域的感兴趣区域(ROI)进行提取;4)在点扩展函数(PSF)梯度方向实施定位的非迭代单分子定位算法[26]。目前,WindSTORM提供了两种方式进行超分辨数据处理,一种是基于MATLAB只采用中央处理器(CPU)进行数据处理,另一种是采用图形处理器(GPU)对CUDA(compute unified device architecture)编译后的可执行文件进行数据处理[23]。但目前的GPU版本只能处理raw格式的数据,而普通科研级相机(例如Photometrics的Andor)均不能直接导出该格式,需花费大量额外的时间进行格式转换。而且,采用英伟达GeForce GT 730、Quadro M4000、Quadro K2000和Quadro P5000这四种显卡对WindSTORM(GPU)进行测试后发现,其在Quadro P5000下无法运行,在GeForce GT 730、Quadro M4000和Quadro K2000下得到的结果与在WindSTORM(CPU)下得到的结果存在较大差异,不具有广泛的稳定适用性。所以,本文使用WindSTORM(CPU)进行算法的对比以及并行优化。
2.2 并行架构
GPU特殊的硬件结构设计使其可以同时运行数千个并发结构线程,因此多被用于数据并行和计算密集的算法中,其处理架构如
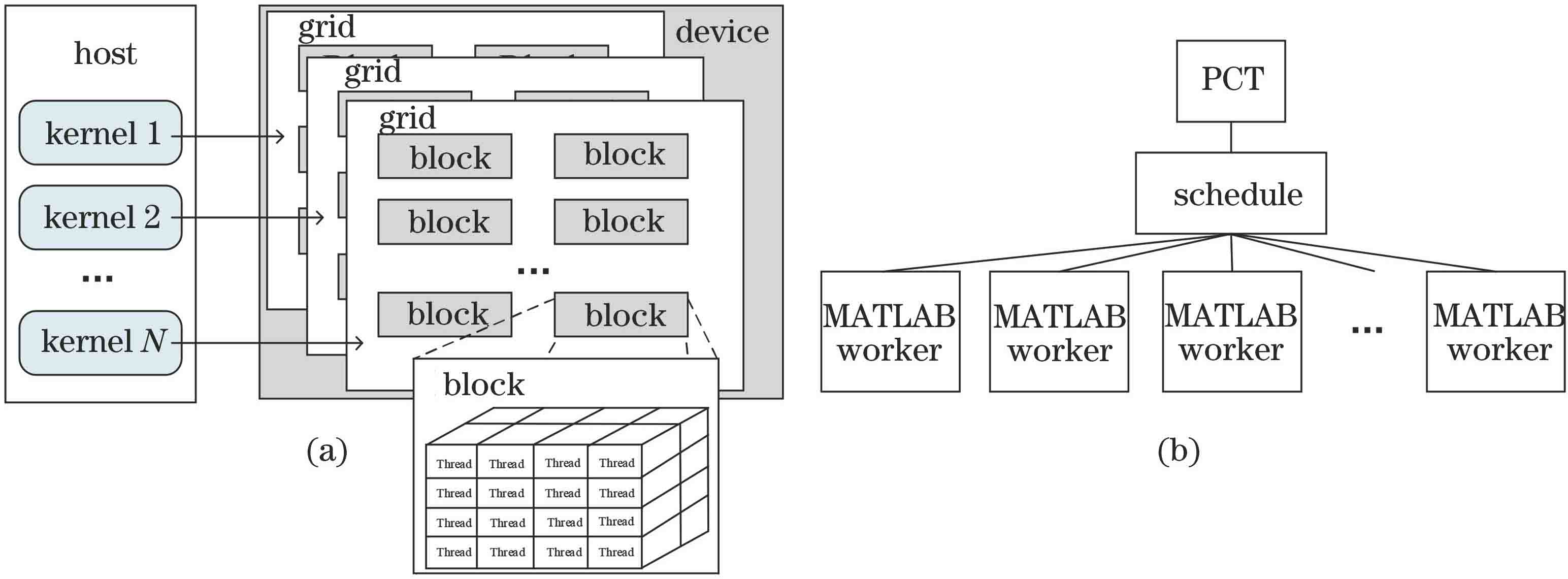
图 1. 架构示意图。(a) GPU架构;(b) MATLAB CPU并行架构
Fig. 1. Schematics of architecture. (a) GPU architecture; (b) MATLAB CPU parallel architecture
2.3 WindSTORM PLUS设计
针对WindSTORM算法的特点,本文将数据处理任务分解为数据预处理、流程控制(根据尺寸选择对应的并行加速方式)、并行计算(背景去除、去卷积频率滤波、梯度方向拟合定位)以及数据后处理等步骤。本文将WindSTORM PLUS设计成如
串行计算部分主要包括数据预处理、流程控制及数据后处理。在数据的预处理中,本文引入开源显微镜环境(OMERO)工具bfmatlab[27],用它替代MATALB图像处理工具箱提供的图像读取函数,这样就可以解决MATALB无法一次性读取4 GB内存以上的图像堆栈的问题。然后,对图像尺寸进行补零调整,以匹配滤波去卷积过程中的截止频率,使WindSTORM PLUS能够处理任意尺寸的数据集。在串行计算的流程控制上,对数据集的并行处理方式是读入后智能选择。在背景去除、去卷积频率滤波和梯度方向拟合定位步骤中,分别采用CPU和GPU对不同尺寸数据集的运行速度进行测试,结果发现:去卷积运算中使用的快速傅里叶变换(FFT)及反变换在处理小尺寸数据集时,CPU的计算速度比GPU更快;在处理大尺寸数据以及其余两个步骤时,GPU的计算速度更快。这是因为在CPU下,FFT的计算时间复杂度为Nlog2N(N为计算点数),CPU下的FFT运算时间随尺寸呈对数增长;而GPU运算的底层使用的是CUDA的cuFFT运算库,其计算时间随尺寸的变化相对平稳。基于此计算分析,本文综合各数据集所有步骤的运行速度,采用读入后智能选择的方式对数据集进行处理,对图像长、宽小于256的小尺寸数据集只使用CPU进行并行计算,对大尺寸数据集择则选择CPU与GPU混合并行计算的模式。在数据后处理中对并行计算结果进行元胞数组数据结构的调整与合并,确保定位点顺序与实际一致,以保证进一步漂移校正等后处理结果的正确性。
并行计算部分主要是算法的计算过程。如
2.4 模拟验证
模拟数据的获取由ImageJ插件ThunderSTORM中的模拟数据生成(generator of simulated data)实现,分别在低密度0.2 emitter/μm-2和高密度1 emitter/μm-2下随机产生成像视野为128×128、256×256、512×512、1024×1024的10000张堆栈数据集,每个图像尺寸下获取5组数据。其余条件均保持一致性,相机模型像素对应物面大小为108 nm,增益值为0.67,基准背景为120,量子效率为95%,电子读取噪声为1.3,发射器模型为高斯模型,半峰全宽(FWHM)为200~350 nm,强度为700~2000,背景平均噪声为50。
本文所有对比结果的数据处理都在惠普Z840工作站上进行,其CPU为Intel(R)-Xeon(R)E5-2643v4,256 GB内存,GPU为NVIDIA Quardra P5000。程序运行环境为MATLAB 2018b。分别选用ThunderSTORM的最小二乘法的高斯拟合(Gauss-WLS)和向量法(Phasor)以及WindSTORM、WindSTORM PLUS进行对比,每秒钟的定位点结果如

图 3. 模拟数据集下不同算法的处理速度对比。(a)低密度模拟数据集;(b)高密度模拟数据集
Fig. 3. Comparison of processing speed of different algorithms using simulated data sets. (a) Data set with low density; (b) data set with high density
本文选用F1分数(F1 score)[28]对上述不同方法下处理模拟数据的准确度进行评价。F1分数是用来评估模型准确度的一个参数,同时兼顾了召回率与准确率。其计算公式为
式中:P为精确率,P=
由
表 1. 模拟数据集下不同算法的准确度评估(F1分数)
Table 1. Accuracy evaluation (F1 score) of different algorithms using simulated data sets%
|
为进一步比较,在高密度下选取128×128×10000的数据集,对常用STORM算法FALCON、Peak Fit和ThunderSTORM中的最大似然估计(MLE)的高斯拟合进行了测试。FALCON每秒钟定位点数为22左右,ThunderSTORM(Gauss-MLE)每秒钟定位点数为129左右,Peak Fit每秒钟定位点数为110左右。FALCON、Peak Fit和ThunderSTORM(Gauss-MLE)这三种算法的处理速度较慢,所以并未作进一步对比。
综合
3 系统及结果分析
3.1 dSTORM成像系统
本文基于easySTORM[24]搭建的STORM成像系统如
3.2 数据采集
本文中的dSTORM样本由帝国理工学院物理系光学组提供。数据采集所用相机为sCMOS(Photometrics Prime BSI,像元尺寸6.5 μm,量子效率95%)。物镜为60倍油镜(Olympus UPlanFLN 60x/1.25 Oil Iris)。采样间隔为108.3 nm。在数据采集过程中,使用的功率约为1 kW/cm2,曝光时间为30 ms,采样频率为33.3 Hz。根据单分子点闪烁密度,设置常规采集张数为5000帧或10000帧,采集时间为2.5 min或5 min左右。
3.3 结果分析
对采集到的Alexa Fluor 647染色微管的图像进行重建,采集10000帧图像,分别用WindSTORM PLUS、ThunderSTORM(Phasor)、ThunderSTORM(Gauss-WLS)定位方法进行处理,重建区域为512×512。

图 5. Alexa Fluor 647染色微管图像的重建结果。(a)明场荧光图像;(b) ThunderSTORM(Phasor)重建图像;(c) ThunderSTORM(Gauss-WLS)重建图像;(d) WindSTORM PLUS重建图像;(e) ROI区域实线的归一化强度拟合; (f)计算的FRC分辨率(R)
Fig. 5. Reconstruction results of microtubule stained by Alexa Fluor 647. (a) Wide-field fluorescence image; (b) super-resolution image reconstructed by ThunderSTORM(Phasor); (c) super-resolution image reconstructed by ThunderSTORM(Gauss-WLS); (d) super-resolution image reconstructed by WindSTORM PLUS; (e) normalized intensity fitting of the line of ROI; (f) calculated FRC resolution (R)
为进一步验证大视野范围成像及定位结果,使用Phalloidin atto 647对人胚胎肾细胞(HEK-293)的肌动蛋白进行染色,然后进行STORM成像,重建区域为1024×1024,重建帧数为5000。分别采用ThunderSTORM(Phasor)、ThunderSTORM(Gauss-WLS)、WindSTORM PLUS算法进行重组处理,得到的超分辨图像分别如

图 6. 人胚胎肾细胞(HEK-293)肌动蛋白的超分辨图像。(a)明场荧光图像;(b) ThunderSTORM(Phasor)重建图像;(c) ThunderSTORM(Gauss-WLS)重建图像;(d) WindSTORM PLUS重建图像
Fig. 6. Super-resolution image of HEK-293 actin. (a) Wide-field fluorescence image; (b) super-resolution image reconstructed using ThunderSTORM (Phasor); (c) super-resolution image reconstructed using ThunderSTORM (Gauss-WLS); (d) super-resolution image reconstructed using WindSTORM PLUS
4 结论
本文构造了开源WindSTORM PLUS算法,该算法采用并行计算大幅提升了重组速度,并可适用于STORM任意尺寸数据集的处理。通过对不同尺寸和不同密度的模拟数据进行处理后发现,在大数据集下,WindSTORM PLUS相较WindSTORM和ThunderSTORM(Gauss-WLS),定位速度提高了1000%,相较ThunderSTORM(Phasor)提高了50%。此外,本文采用搭建的easySTORM成像系统对实际样本进行了成像和数据处理,在512×512×10000和1024×1024×5000的数据集下,WindSTORM PLUS的耗时只有WindSTORM和ThunderSTORM(Gauss-WLS)的9%,ThunderSTORM(Phasor)的70%,且利用FRC计算的分辨率略有提高。本文构建的WindSTORM PLUS算法为超分辨图像处理提供了一个全新的高速处理方案,其程序已发布到开源社区(https:∥github.com/Northstar1994/WindSTORM_PLUS)。可以相信,STORM数据处理速度还可通过CUDA编程编译的MEX文件加强并行计算以及引入深度学习等方式得到进一步提高。
[2] Han J J, Kunde YA, Hong-Geller E, et al. Actin restructuring during Salmonella typhimurium infection investigated by confocal and super-resolution microscopy[J]. Journal of Biomedical Optics, 2014, 19(1): 016011.
[3] 付芸, 王天乐, 赵森. 超分辨光学显微的成像原理及应用进展[J]. 激光与光电子学进展, 2019, 56(24): 240002.
[4] 林丹樱, 屈军乐. 超分辨成像及超分辨关联显微技术研究进展[J]. 物理学报, 2017, 66(14): 148703.
Lin D Y, Qu J L. Recent progress on super-resolution imaging and correlative super-resolution microscopy[J]. Acta Physica Sinica, 2017, 66(14): 148703.
[5] Rust M J, Bates M, Zhuang X W. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM)[J]. Nature Methods, 2006, 3(10): 793-796.
[6] Gustafsson M G L. Surpassing the lateral resolution limit by a factor of two using structured illumination microscopy[J]. Journal of Microscopy, 2000, 198(2): 82-87.
[7] Westphal V, Rizzoli S O, Lauterbach M A, et al. Video-rate far-field optical nanoscopy dissects synaptic vesicle movement[J]. Science, 2008, 320(5873): 246-249.
[8] Heilemann M. Van de Linde S, Schüttpelz M, et al. Subdiffraction-resolution fluorescence imaging with conventional fluorescent probes[J]. Angewandte Chemie International Edition, 2008, 47(33): 6172-6176.
[10] Huang F. Hartwich T M P,Rivera-Molina F E, et al. Video-rate nanoscopy using sCMOS camera-specific single-molecule localization algorithms[J]. Nature Methods, 2013, 10(7): 653-658.
[11] 桂丹, 商明涛, 黄振立. 基于sCMOS相机的超分辨定位成像技术[J]. 中国激光, 2018, 45(2): 0207016.
[13] Huang F, Schwartz S L, Byars J M, et al. Simultaneous multiple-emitter fitting for single molecule super-resolution imaging[J]. Biomedical Optics Express, 2011, 2(5): 1377-1393.
[14] Nandy K, Chellappa R, Kumar A, et al. Segmentation of nuclei from 3D microscopy images of tissue via graphcut optimization[J]. IEEE Journal of Selected Topics in Signal Processing, 2016, 10(1): 140-150.
[16] Holden S J, Uphoff S, Kapanidis A N. DAOSTORM: an algorithm for high- density super-resolution microscopy[J]. Nature Methods, 2011, 8(4): 279-280.
[17] Min J H, Vonesch C, Kirshner H, et al. FALCON: fast and unbiased reconstruction of high-density super-resolution microscopy data[J]. Scientific Reports, 2015, 4: 4577.
[18] Smith C S, Joseph N, Rieger B, et al. Fast, single-molecule localization that achieves theoretically minimum uncertainty[J]. Nature Methods, 2010, 7(5): 373-375.
[19] Brede N, Lakadamyali M. GraspJ: an open source, real-time analysis package for super-resolution imaging[J]. Optical Nanoscopy, 2012, 1(1): 11.
[21] Martens K J A, Bader A N, Baas S, et al. Phasor based single-molecule localization microscopy in 3D (pSMLM-3D): an algorithm for MHz localization rates using standard CPUs[J]. The Journal of Chemical Physics, 2018, 148(12): 123311.
[22] Sage D, Kirshner H, Pengo T, et al. Quantitative evaluation of software packages for single-molecule localization microscopy[J]. Nature Methods, 2015, 12(8): 717-724.
[23] Sage D, Pham T A, Babcock H, et al. Super-resolution fight club: assessment of 2D and 3D single-molecule localization microscopy software[J]. Nature Methods, 2019, 16(5): 387-395.
[24] Ma H Q, Xu J Q. 5(4): eaaw0683[J]. Liu Y. WindSTORM: robust online image processing for high-throughput nanoscopy. Science Advances, 2019.
[25] Kwakwa K, Savell A, Davies T, et al. EasySTORM: a robust, lower-cost approach to localisation and TIRF microscopy[J]. Journal of Biophotonics, 2016, 9(9): 948-957.
[26] Ma H Q, Long F, Zeng S Q, et al. Fast and precise algorithm based on maximum radial symmetry for single molecule localization[J]. Optics Letters, 2012, 37(13): 2481-2483.
[28] SepulvedaJ, Velastin SA. F1 score assesment of Gaussian mixture background subtraction algorithms using the MuHAVi dataset[C]∥6th International Conference on Imaging for Crime Prevention and Detection (ICDP-15), London, UK. [S.l.]: IEEE, 2015: 15382044.
[29] Nieuwenhuizen R P J, Lidke K A, Bates M, et al. Measuring image resolution in optical nanoscopy[J]. Nature Methods, 2013, 10(6): 557-562.
Article Outline
肖文, 吴天琦, 李仁剑, 唐黎, 陈玲玲. 基于并行计算优化的WindSTORM PLUS算法[J]. 中国激光, 2020, 47(6): 0607001. Xiao Wen, Wu Tianqi, Li Renjian, Tang Li, Chen Lingling. WindSTORM PLUS Algorithm with Parallel Computing Optimization[J]. Chinese Journal of Lasers, 2020, 47(6): 0607001.








