生物医学检测中太赫兹光谱技术的算法研究  下载: 2192次特邀综述
下载: 2192次特邀综述
1 引言
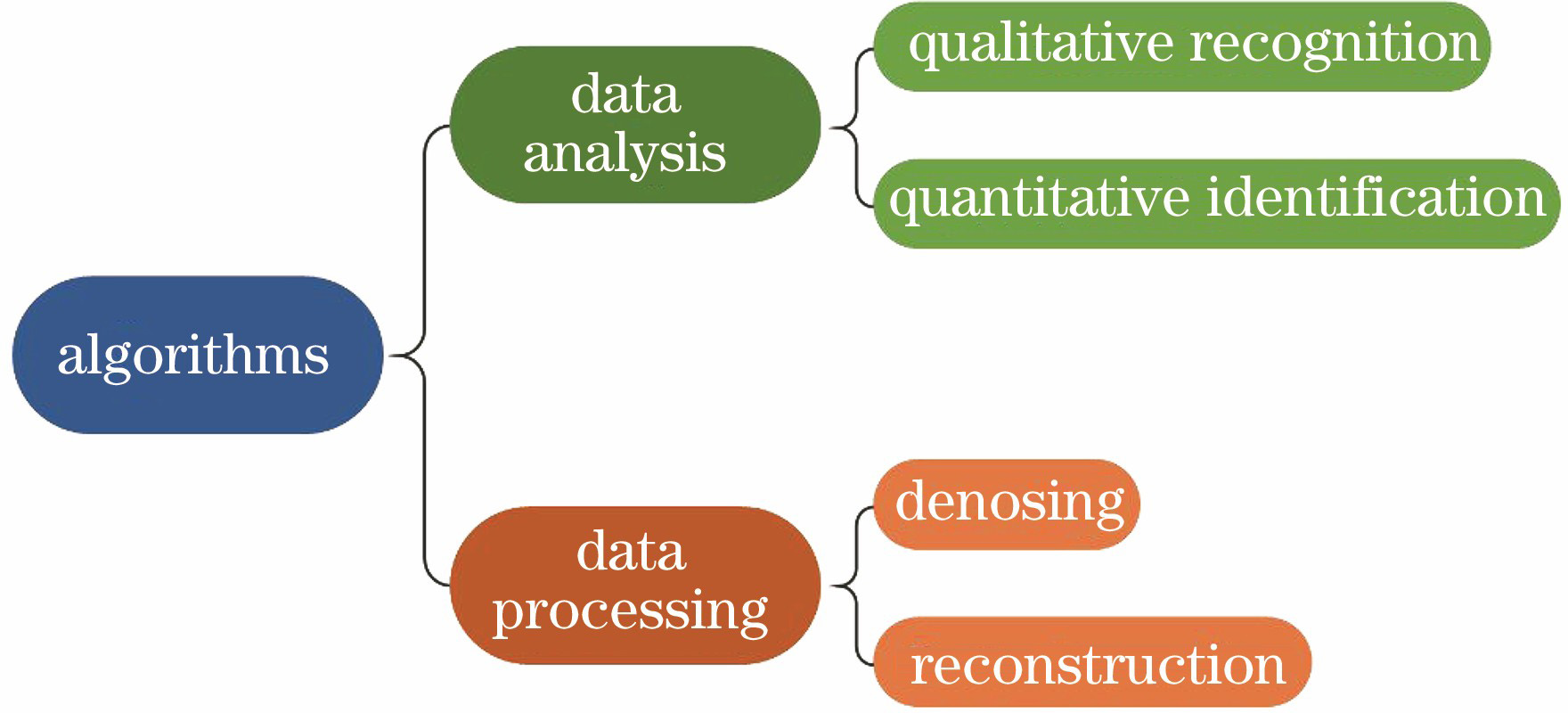
太赫兹(THz) 波位于毫米波区域和红外区域之间,其频率为0.1~10 THz,对应波长为0.3~30 mm[1],兼具毫米波与红外波的特征,并具有非电离性、非侵入性、高穿透性、高分辨率和指纹谱识别等优势,在生物医学领域具有巨大的应用潜力[2-3]。国内外多个研究小组已基于太赫兹光谱技术对多种生物分子进行了识别和分析,包括对不同疾病的生物标记物[4-8]、药物的主要成分[9-13]以及脱氧核糖核酸(DNA)[14-17]进行识别和分析。研究人员除了对纯品的太赫兹光谱进行研究以外,还采用不同的数据分析算法对混合物的太赫兹光谱进行了分析,实现了对混合物样品的定性和定量识别。但是,实际的生物混合物样品中包含水在内的大量的不同成分,这些成分会对太赫兹波产生大量吸收,导致光谱的信噪比(SNR)较差,光谱分析结果的误差较大[18]。因此,也有研究人员以不同的数据处理算法作为辅助手段,通过提升光谱的信噪比来提高分析的准确率。本文对近五年来上述分析和处理算法进行了归纳,如
在定性和定量分析算法的研究方面,人们主要通过机器学习或线性回归算法来实现生物样品的定性、定量识别,他们主要使用最小二乘回归、支持向量机(SVM)等算法对光谱数据进行分析建模,从而做到对未知样本的定性、定量预测。但是,当光谱的信噪比较差时,会出现建模误差,从而使最终的预测准确率大幅下降。因此,有研究小组在应用这些分析算法前,先使用降噪或重构算法对光谱数据进行预处理,以提高数据的信噪比。他们主要使用的算法有小波变换、主成分分析(PCA)和匹配算法等。经这些算法处理后的数据相比原始数据具有更高的信噪比,可以提升最终分析结果的准确率。
本文总结了近五年来这些算法在生物医学领域的应用情况,并归纳了它们的优缺点。
2 应用于数据分析的定性和定量分析算法
近五年来,有许多研究人员采用不同的算法对不同的物质进行了定性和定量分析,本章将对目前所用的几种主流算法一一进行介绍。
偏最小二乘(PLS)回归是一种利用线性多元模型将两个数据矩阵X和Y进行关联的算法[19],它可将目标物质的浓度与混合物的光谱建立关联,从而实现混合物中各成分的定性、定量预测。目前,大部分研究工作都是基于PLS算法实现样本定性和定量分析的。
2016年,Liu等[20]将果糖浆含量不同(果糖浆质量分数为10%~100%)的蜂蜜作为样本,将样本光谱导入PLS回归算法中建立模型,该模型对果糖浆含量预测的均方根误差(RMSEP)可达0.108。Lu等[21]以粟米为基质制备了谷氨酰胺和谷氨酸的二元混合物(两种物质的质量分数均为0~13%),然后采用区间PLS回归算法对这两种氨基酸进行定量测试;测试结果表明,谷氨酰胺和谷氨酸的RMSEP均为0.39,相关系数(R)均为0.99。2017年,Yuan等[22]制备了以聚乙烯为基底的诺氟沙星药片,并通过PLS回归进行定性和定量分析;分析结果表明,所建立模型的R和均方根误差(RMSE)分别达到0.9908和0.0481,检测限为10% (质量分数)。Wang等[23]采用PLS算法对不同品种苜蓿的太赫兹光谱进行了识别,识别结果显示,该算法对8种苜蓿分类的RMSE为1.0596。Liu[24]采用偏最小二乘判别分析(PLS-DA)对转基因和非转基因玉米油的光谱进行了分类,20组样本的验证实验说明,该算法的分类准确率(实验结果与实际结果的比值)为98.7%。da Silva等[25]采用PLS算法对甲苯咪唑(MBZ)的三种晶型(晶型A、B、C)进行了定量分析,回归模型获得的检测限(质量分数)分别为2.7%~4.3%、2.9%~4.0%和2.4%~3.1%,三种晶型的RMSEP分别为1.5%、1.2%和1.8%。Nie等[26]基于核函数偏最小二乘(KPLS)算法对菜籽叶中的水含量进行了测定,得到了基于太赫兹透射光谱和太赫兹吸收光谱所建模型的预测结果,如
![使用KPLS模型进行水含量预测的校准和验证结果[26]。(a)根据透射光谱建模;(b)根据吸收光谱建模](/richHtml/gxxb/2021/41/1/0130001/img_2.jpg)
图 2. 使用KPLS模型进行水含量预测的校准和验证结果[26]。(a)根据透射光谱建模;(b)根据吸收光谱建模
Fig. 2. Calibration and validation results for water content prediction using KPLS models[26].(a) Established by the transmission spectra; (b) established by the absorption spectra
部分成分分析对效率、精度的要求很高,因此,部分研究小组提出使用支持向量机(SVM)进行分析。SVM是一种机器学习算法,它在解决小样本、非线性和高维模式识别问题时具有独特优势[30],可用于光谱数据的分类识别;同时,基于SVM的回归算法——支持向量回归(SVR)还可实现对物质的定量检测。SVM算法中存在不同的核函数,用于对当前维度下无法实现线性区分的数据进行升维,从而使数据在其他维度上线性可分[31]。对于光谱数据而言,当模型中包含有大量不同物质的样本时,不同物质的部分特征峰可能存在叠加,导致这些物质的特征峰无法被线性区分。因此,核函数能通过对光谱升维来有效区分这些光谱,实现对光谱的定性和定量分析。
目前,SVM/SVR也是一种用于定性/定量分析混合物太赫兹光谱的主流算法。2016年,Li等[32]基于自适应粒子群优化支持向量机(APSO-SVM)算法对转基因棉花种子光谱进行了分类,三种转基因棉花种子的165个样品的分析结果表明,模型的总识别率高达97.3%。2017年,Qin等[33]制备了多菌灵和聚乙烯的混合物以及多菌灵和米粉的混合物,并基于SVR定量分析了混合物中多菌灵的含量,两种混合物SVR模型的RMSEP分别为0.0200和0.0188,R分别为0.9972和0.9978,检测限(质量分数)为2%。2018年,Sun等[34]测试了牛血清白蛋白(BSA)薄膜的光谱,并用SVR分析了质量浓度为0.5~35 mg/mL的BSA薄膜的光谱数据,分析结果如
![排除交叉验证(LOOCV)-SVR对0.5~35 mg/mL BSA薄膜的预测[34]。(a) 实际浓度和预测浓度的分布,横轴的有效值为1~135,代表135个光谱测量值;(b) 预测浓度与实际浓度的关系](/richHtml/gxxb/2021/41/1/0130001/img_3.jpg)
图 3. 排除交叉验证(LOOCV)-SVR对0.5~35 mg/mL BSA薄膜的预测[34]。(a) 实际浓度和预测浓度的分布,横轴的有效值为1~135,代表135 个光谱测量值;(b) 预测浓度与实际浓度的关系
Fig. 3. LOOCV-SVR prediction for BSA with various concentrations in the range from 0.5 to 35 mg/mL. (a) Distributions of actual and predicted concentrations, the valid value of horizontal axis is from 1 to 135 and represents 135 spectral measurement values; (b) actual concentrations against predicted concentrations
相比PLS算法,SVM算法可以识别更低浓度的样品[39]。综合上述工作可以发现,SVM算法可以达到的检测限为0.05%(质量分数,测试对象为苯甲酸),相比PLS算法的0.8%(质量分数,测试对象为一水乳糖)提高了一个数量级。但是,由于SVM算法在建模时需要设置参数及核函数,相关的寻优过程是必不可少的,因此计算时间要远超PLS算法。
除此之外,还有些研究采用了其他算法。比如:Peng等[40]制备了包含两种神经递质——γ-氨基丁酸(GABA)和L-谷氨酸(L-Glu)以及两种典型代谢物——肌醇(D-MI)和肌酸(CMH)的四元混合物,然后采用最小二乘法(LSM)预测了4种物质的含量,并拟合得到了混合物的光谱,如
![L-Glu、D-MI、GABA与CMH混合药片的光谱[40]。(a)混合药片的实验结果;(b)实验结果与计算结果的对比](/richHtml/gxxb/2021/41/1/0130001/img_4.jpg)
图 4. L-Glu、D-MI、GABA与CMH混合药片的光谱[40]。(a)混合药片的实验结果;(b)实验结果与计算结果的对比
Fig. 4. The spectra of mixture made by L-Glu, D-MI, GABA and CMH[40].(a) Experimental results of mixture; (b) comparison between experiment result and calculation result
综合上述研究工作可以发现,对于不同的物质,其适应的最佳算法也不同。因此,对于此类基于算法的定性和定量分析测试,必须要综合考虑各种算法,并从中选取效果最佳的算法。但是,此类分析算法一般需要具有较高信噪比的光谱用于训练模型,当光谱的信噪比较差时,会导致训练出的模型准确度大幅下降。因此,大部分研究会在分析前使用各类数据处理算法来优化光谱数据,间接提高分析算法的准确性。
3 应用于数据处理的降噪重构算法
对于实际的生物混合物样品,由于它会受到包括水在内的不同物质的干扰,光谱的信噪比较差,因此,对于此类信噪比较低的光谱,在应用分析算法前需要首先使用处理算法来提高其光谱的信噪比。鉴于此,本节将对目前使用的几种主流数据处理算法逐一进行介绍。
遗传算法是一种借鉴生物自然选择和自然遗传机制的随机搜索算法,该算法通过迭代从群体中选取较优的个体[44]。目前,有些研究人员利用遗传算法来选取建立算法模型的最优变量,目的是提升模型识别的准确率。
2016年,Yin等[45]基于遗传算法结合PLS回归对五种食用油(花生油、玉米油、胡椒油、芝麻油和葵花籽油)进行了光谱分类,与普通PLS算法相比,他们采用的算法的识别准确率高达100%。Li[46]测试了12个由谷氨酰胺和组氨酸混合而成的样品中的谷氨酰胺含量,采用遗传算法选出与浓度相关的波长用于定量分析;分析结果表明,遗传算法对12个混合样品的定量分析误差在6%以下,标准偏差为0.0344。Qin等[47]测得了4种不同转基因棉花种子的光谱,采用PCA算法提取前12个主成分,然后应用多种群遗传算法(MPGA)结合SVM选取最优变量建立模型;对于4种棉花共192例样本,所建模型的识别准确率高达99%。2018年,Liu等[48]基于LS-SVM结合遗传算法对4种不同的特级初榨橄榄油进行光谱分类,对于80个预测集样本,模型的预测准确率为96.25%。
遗传算法能够从光谱数据中选出与待测物质相关的特征吸收峰信息,从而在建立模型时排除影响模型性能的其他频段的无效信息,提升模型的准确性。但是,遗传算法仅能从数据中直接提取出有效信息进行分析,不会对这些信息进行转换,因此不能提升区分度。
主成分分析(PCA)能够将一组高维度数据重构为主成分的新变量,这些变量是原始数据的线性组合,且第一个主成分具有最大的方差[49]。由于PCA算法能大幅降低太赫兹光谱的维度,并可将不同样本光谱数据的差异最大化,因此可以提高定性和定量分析的精度。目前,许多定性、定量研究工作都在数据分析前使用PCA算法对数据进行重构。
2016年,Nie等[50]使用PCA算法从大豆的光谱中提取了8个主成分,将主成分输入至反向传播神经网络(BPNN)算法中用于识别转基因大豆,识别的累计方差为97.582%。Zhan等[51]使用PCA算法对食用油和污油的光谱进行降维,其中第一主成分(PC1)识别了97.4%的光谱特征,随后采用SVM算法对污油实现了100%的识别率。Ge等[39]基于PCA-SVM算法对黄曲霉毒素B1的乙腈溶液进行了定量分析,分析结果表明,当黄曲霉毒素B1的质量浓度为1~50 μg/L时,模型的预测准确率可达93.75%,相比直接使用SVM算法提高了8.75%。Zhang等[52]测试了三种不同中草药(白英、龙葵和马兜铃草)的光谱,采用PCA降低原始光谱的维度,并通过RF算法进行分类,模型对100个测试样本的分类准确性高达99%。2017年,Zou等[53]采用PCA算法对正常和实验性自身免疫性脑脊髓炎(EAE)猴脑组织的太赫兹光谱进行了区分,结果如
![实验性自身免疫性脑脊髓炎(EAE)和正常猴脑组织前两个主成分的评分图[53]](/richHtml/gxxb/2021/41/1/0130001/img_5.jpg)
图 5. 实验性自身免疫性脑脊髓炎(EAE)和正常猴脑组织前两个主成分的评分图[53]
Fig. 5. Score plot of the first two principle components for the EAE and normal monkey brain tissues[53]
PCA不仅可以实现对光谱数据的降维,还能够重构光谱数据,将目标物质的特征更直观地表现出来。但是,在光谱分析工作中,通常选用前两个或前三个主成分值进行分析,这些主成分值能表征95%以上的光谱信息,但并不能表征所有的光谱信息,因此会有部分信息丢失而产生细微误差。
降噪重构算法不仅能以最少的维度表征光谱的有效信息,将不同物质的光谱差异最大化,而且能够大幅压缩光谱信息,加快识别效率。但是,此类算法无法实现对光谱的降噪。当光谱噪声过大时,这类算法会将光谱的噪声识别为光谱特征并进行提取,导致最终结果产生误差。因此,对于低信噪比光谱进行分析时,各研究小组通常使用不同的数据降噪算法来提升光谱的信噪比。
小波变换通常是将光谱信号拆解为对应不同频段的组分,进而去除代表噪声的高频组分,最后采用小波逆变换重组光谱信号。与传统的小窗傅里叶变换降噪相比,小波变换更适用于诸如太赫兹时域信号之类的瞬时变化的非平稳信号[59]。
2018年,Zhang等[60]测得了6-苄基氨基嘌呤(6-BA)、多效唑(PBZ)和马来酰肼(MH)这三种植物生长调节剂的太赫兹光谱,然后基于“sym4”小波函数和四层小波对光谱进行分解,处理后的光谱实现了更高的信噪比(6-BA的信噪比为40.22,PBZ的信噪比37.73,MH的信噪比34.83)和更低的RMSE(6-BA的RMSE为0.41,PBZ的RMSE为0.40,MH的RMSE为0.54)。Peng等[61]测得了存在于脑胶质瘤中的混合物(7种物质)的光谱,然后基于小波变换和多项式拟合基线校正对光谱进行去噪处理并去除基线,结果如
![10个混合物样品的太赫兹吸收光谱[61]。(a)原始光谱;(b)小波变换后的光谱;(c)通过多项式拟合进行基线校正后的光谱](/richHtml/gxxb/2021/41/1/0130001/img_6.jpg)
图 6. 10个混合物样品的太赫兹吸收光谱[61]。(a)原始光谱;(b)小波变换后的光谱;(c)通过多项式拟合进行基线校正后的光谱
Fig. 6. Terahertz absorption spectra of ten mixture samples[61]; (a) Original spectra; (b) spectra after wavelet transform; (c) spectra after baseline correction by polynomial fitting
目前,最常用的Savitzky-Golay平滑是将光谱进行整体平滑,因而会将光谱中物质的特征吸收和噪声一起去除。与常用的Savitzky-Golay平滑去噪相比,小波变换能在去除光谱噪声的同时保留光谱的有效信息。但是,小波变换中存在不同的小波函数,需要根据数据的实际情况选用合适的小波,错误选择将会导致光谱的有效信息大幅丢失。
除了上述主流方法,也有许多研究使用了其他算法进行降噪或重构光谱数据。如:2017年,Zhang等[63]测得了L-谷氨酸、L-谷氨酰胺和L-酪氨酸三元混合物的光谱,研究了多元散射校正(MSC)、Savitzky-Golay(S-G)平滑、一阶导数和小波变换等预处理算法,并采用PLS与SVM算法对三种组分的含量进行了定量分析;其中,将MSC与SVM算法相结合的方法具有最佳效果,对质量分数为3.33%~20%的样品进行识别时,该方法对L-谷氨酸、L-谷氨酰胺和L-酪氨酸识别的相关系数分别可达0.9993、0.9997和0.9994。Türker-Kaya等[64]测量了转基因与非转基因水稻种子的光谱,然后采采稀疏表示(SR)算法提取光谱特征,并采用RF算法建立识别模型,该模型对于60例样本的识别结果显示,识别准确率可达95%。2018年,Liu等[65]采集了肝脏肿瘤组织以及肝脏正常组织的太赫兹光谱,然后使用局部保持投影(LPPs)对光谱进行压缩降维,并使用概率神经网络(PNN)进行肿瘤识别,该方法对测试集中的20例样本实现了100%的识别准确率。2019年,Liu等[66]采用太赫兹光谱法快速测定了污染大豆油中致癌物质黄曲霉毒素B-1(AFB)的含量;他们将t-SNE作为预处理方法,采用BPNN进行定量分析,模型的R为0.9948,RMSEP为0.7124 μg/kg,检测限可达1 μg/kg。Huang等[67]基于最大信息系数(MIC)提取光谱特征,并将其用于小鼠肝损伤的识别;他们首先使用统计技术箱线图剔除存在数据异常的光谱,然采用MIC提取光谱特征,最后使用RF和AdaBoost算法进行分类,识别率均为92%。2020年,Huang等[68]测试了质量浓度为0.2~50 mg/mL的糖蛋白溶液,发现吸收系数与浓度之间存在显著的非线性关系;他们通过复合多尺度熵(CMSE)方法获得了特征,并通过K均值算法聚类实现了对糖蛋白溶液中糖蛋白含量的定量识别,准确率最高可达90%。
此类信号处理的算法均通过提取有效信息或消除无关信息来提升光谱数据的信噪比,但是这些算法通常不具备分析功能,因此需要结合分析算法进行定性、定量分析。此外,这些算法通常包含各种参数,需要根据自身数据情况进行调整。设置参数错误可能会导致样品信息丢失,并最终导致光谱识别错误。
4 结束语
本文对目前生物医学领域中基于太赫兹光谱技术的各种算法的研究工作进行了介绍,这些算法包括两部分:应用于数据分析的定性和定量算法以及应用于数据处理的降噪和重构算法。
应用于数据分析的定性和定量算法通过使用各类回归算法或机器学习算法对光谱进行识别来实现对样本的定性和定量分析,但此类算法通常需要高信噪比的光谱来建立模型;而在生物医学领域,样本通常包含水在内的各种物质,光谱的信噪比较差,导致所建模型的识别准确率较低。因此,在应用这类分析算法前,应先采用降噪重构算法从光谱中提取可用于建立模型的关键信息,同时消除噪声信号,以提高光谱的信噪比;之后再采用分析算法对处理后的信号建立识别模型,以有效提高识别准确率。但是,此类算法一般需要结合样本实际情况设置各种参数,参数的不当设置会导致样本中的有效数据丢失。
通过介绍这些研究工作,本研究团队希望读者可以了解到目前生物医学领域太赫兹检测过程中结合算法的优势。此外,在目前的分析算法研究中,用于模型训练的样本和预测样本均为同一类型的样本,它们的组分一致,仅在含量上有所区别;然而在实际情况中,样本中通常会包含一些未知成分。因此,未来的太赫兹算法研究应着重于使用不同的样本(除了含有目标组分以外,各个样本中含有不同种类的其他组分),以配合太赫兹仪器在临床医学中快速、准确地开展疾病检测工作。
[1] Pickwell E, Wallace V P. Biomedical applications of terahertz technology[J]. Journal of Physics D: Applied Physics, 2006, 39(17): R301-R310.
[2] Danciu M, Alexa-Stratulat T, Stefanescu C, et al. Terahertz spectroscopy and imaging: a cutting-edge method for diagnosing digestive cancers[J]. Materials, 2019, 12(9): 1519.
[3] Peng Y, Zhu Y M, Gu M, et al. Terahertz spatial sampling with subwavelength accuracy[J]. Light: Science & Applications, 2019, 8: 72.
[4] Li T, Ma H Y, Peng Y, et al. Gaussian numerical analysis and terahertz spectroscopic measurement of homocysteine[J]. Biomedical Optics Express, 2018, 9(11): 5467-5476.
[5] Chen W Q, Peng Y, Jiang X K, et al. Isomers identification of 2-hydroxyglutarate acid disodium salt (2HG) by terahertz time-domain spectroscopy[J]. Scientific Reports, 2017, 7(1): 12166.
[6] Altan H, Ozek N S, Gok S, et al. Monitoring of tryptophan as a biomarker for cancerous cells in terahertz (THz) sensing[J]. Proceedings of SPIE, 2016, 9703: 97030X.
[7] Joseph C S, Yaroslavsky A N. Munir Al-Arashi M D, et al. Terahertz spectroscopy of intrinsic biomarkers for non-melanoma skin cancer[J]. Proceedings of SPIE, 2009, 7215: 72150I.
[8] Wang L P, Wu X, Peng Y, et al. Quantitative analysis of homocysteine in liquid by terahertz spectroscopy[J]. Biomedical Optics Express, 2020, 11(5): 2570-2577.
[9] Zeitler J A, Kogermann K, Rantanen J, et al. Drug hydrate systems and dehydration processes studied by terahertz pulsed spectroscopy[J]. International Journal of Pharmaceutics, 2007, 334(1/2): 78-84.
[10] Kawase K, Ogawa Y, Watanabe Y, et al. Non-destructive terahertz imaging of illicit drugs using spectral fingerprints[J]. Optics Express, 2003, 11(20): 2549-2554.
[11] Sibik J, Löbmann K, Rades T, et al. Predicting crystallization of amorphous drugs with terahertz spectroscopy[J]. Molecular Pharmaceutics, 2015, 12(8): 3062-3068.
[13] Taday P F, Bradley I V, Arnone D D, et al. Using terahertz pulse spectroscopy to study the crystalline structure of a drug: a case study of the polymorphs of ranitidine hydrochloride[J]. Journal of Pharmaceutical Sciences, 2003, 92(4): 831-838.
[14] Fischer B M, Walther M, Uhd Jepsen P. Far-infrared vibrational modes of DNA components studied by terahertz time-domain spectroscopy[J]. Physics in Medicine and Biology, 2002, 47(21): 3807-3814.
[15] Brucherseifer M, Nagel M, Haring Bolivar P, et al. Label-free probing of the binding state of DNA by time-domain terahertz sensing[J]. Applied Physics Letters, 2000, 77(24): 4049-4051.
[16] Markelz A G, Roitberg A, Heilweil E J. Pulsed terahertz spectroscopy of DNA, bovine serum albumin and collagen between 0.1 and 2.0 THz[J]. Chemical Physics Letters, 2000, 320(1/2): 42-48.
[17] Cheon H, Yang H J, Lee S H, et al. Terahertz molecular resonance of cancer DNA[J]. Scientific Reports, 2016, 6: 37103.
[18] Peng Y, Shi C J, Zhu Y M, et al. Terahertz spectroscopy in biomedical field: a review on signal-to-noise ratio improvement[J]. Photoni X, 2020, 1: 12.
[19] Wold S, Sjöström M, Eriksson L. PLS-regression: a basic tool of chemometrics[J]. Chemometrics and Intelligent Laboratory Systems, 2001, 58(2): 109-130.
[20] Liu W, Zhang Y Y, Han D H. Feasibility study of determination of high-fructose syrup content of Acacia honey by terahertz technique[J]. Proceedings of SPIE, 2016, 1003: 100300J.
[21] Lu S H, Zhang X, Zhang Z Y, et al. Quantitative measurements of binary amino acids mixtures in yellow foxtail millet by terahertz time domain spectroscopy[J]. Food Chemistry, 2016, 211: 494-501.
[22] Yuan L, Bin L. Preliminary study on qualitative and quantitative detection of norfloxacin based on terahertz spectroscopy[J]. International Journal of Agricultural and Biological Engineering, 2017, 10(5): 262-268.
[23] Wang F, Guo S. The qualitative identification of different alfalfa breed in same forage series by the terahertz spectroscopy[J]. Proceedings of SPIE, 2017, 1024: 102441R.
[24] Liu J J. Terahertz spectroscopy and chemometrics classification of transgenic corn oil from corn edible oil[J]. Microwave and Optical Technology Letters, 2017, 59(3): 654-658.
[25] da Silva V H, Vieira F S, Rohwedder J J R, et al. Multivariate quantification of mebendazole polymorphs by terahertz time domain spectroscopy (THZ-TDS)[J]. Analyst, 2017, 142(9): 1519-1524.
[26] Nie P C, Qu F F, Lin L, et al. Detection of water content in rapeseed leaves using terahertz spectroscopy[J]. Sensors, 2017, 17(12): 2830.
[27] Liu W, Zhang Y Y, Yang S, et al. Terahertz time-domain attenuated total reflection spectroscopy applied to the rapid discrimination of the botanical origin of honeys[J]. Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy, 2018, 196: 123-130.
[28] Warnecke S, Wu J X, Rinnan Å, et al. Quantifying crystalline α-lactose monohydrate in amorphous lactose using terahertz time domain spectroscopy and near infrared spectroscopy[J]. Vibrational Spectroscopy, 2019, 102: 39-46.
[29] Lian F Y, Ge H Y, Ju X J, et al. Quantitative analysis of trans fatty acids in cooked soybean oil using terahertz spectrum[J]. Journal of Applied Spectroscopy, 2019, 86(5): 917-924.
[30] 丁世飞, 齐丙娟, 谭红艳. 支持向量机理论与算法研究综述[J]. 电子科技大学学报, 2011, 40(1): 2-10.
Ding S F, Qi B J, Tan H Y. An overview on theory and algorithm of support vector machines[J]. Journal of University of Electronic Science and Technology of China, 2011, 40(1): 2-10.
[31] Noble W S. What is a support vector machine?[J]. Nature Biotechnology, 2006, 24(12): 1565-1567.
[32] Li T J, Liu J J, Shao G F, et al. A novel THz spectroscopy recognition method for transgenic organisms based on APSO combined with SVM[J]. Optics and Spectroscopy, 2016, 120(4): 660-665.
[33] Qin B Y, Li Z, Luo Z H, et al. Feasibility of terahertz time-domain spectroscopy to detect carbendazim mixtures wrapped in paper[J]. Journal of Spectroscopy, 2017, 2017: 1-8.
[34] Sun Y W, Du P J, Lu X X, et al. Quantitative characterization of bovine serum albumin thin-films using terahertz spectroscopy and machine learning methods[J]. Biomedical Optics Express, 2018, 9(7): 2917-2929.
[35] Yan L, Liu C H, Qu H, et al. Discrimination and measurements of three flavonols with similar structure using terahertz spectroscopy and chemometrics[J]. Journal of Infrared, Millimeter, and Terahertz Waves, 2018, 39(5): 492-504.
[37] Sun X D, Zhu K, Liu J B, et al. Terahertz spectroscopy determination of benzoic acid additive in wheat flour by machine learning[J]. Journal of Infrared, Millimeter, and Terahertz Waves, 2019, 40(4): 466-475.
[38] Guan A, Chao Y. Quantitative analysis of alum based on terahertz time-domain spectroscopy technology and support vector machine[J]. Optik, 2019, 193: 163017.
[39] Ge H Y, Jiang Y Y, Lian F Y, et al. Quantitative determination of aflatoxin B1 concentration in acetonitrile by chemometric methods using terahertz spectroscopy[J]. Food Chemistry, 2016, 209: 286-292.
[40] Peng Y, Yuan X R, Zou X, et al. Terahertz identification and quantification of neurotransmitter and neurotrophy mixture[J]. Biomedical Optics Express, 2016, 7(11): 4472-4479.
[41] Liu J, Luo J, Li P, et al. Detection of transgenic cotton using THz spectroscopy and weighted discriminate analysis[J]. Journal of Applied Spectroscopy, 2017, 84(2): 346-350.
[42] Long Y, Li B, Liu H. Analysis of fluoroquinolones antibiotic residue in feed matrices using terahertz spectroscopy[J]. Applied Optics, 2018, 57(3): 544-550.
[43] Liu W, Liu C H, Hu X H, et al. Application of terahertz spectroscopy imaging for discrimination of transgenic rice seeds with chemometrics[J]. Food Chemistry, 2016, 210: 415-421.
[44] 马永杰, 云文霞. 遗传算法研究进展[J]. 计算机应用研究, 2012, 29(4): 1201-1206, 1210.
Ma Y J, Yun W X. Research progress of genetic algorithm[J]. Application Research of Computers, 2012, 29(4): 1201-1206, 1210.
[45] Yin M, Tang S F, Tong M M. Identification of edible oils using terahertz spectroscopy combined with genetic algorithm and partial least squares discriminant analysis[J]. Analytical Methods, 2016, 8(13): 2794-2798.
[46] Li Z. Wavelength selection for quantitative analysis in terahertz spectroscopy using a genetic algorithm[J]. IEEE Transactions on Terahertz Science and Technology, 2016, 6(5): 658-663.
[47] Qin B Y, Li Z, Chen T, et al. Identification of genetically modified cotton seeds by terahertz spectroscopy with MPGA-SVM[J]. Optik, 2017, 142: 576-582.
[48] Liu W, Liu C H, Yu J J, et al. Discrimination of geographical origin of extra virgin olive oils using terahertz spectroscopy combined with chemometrics[J]. Food Chemistry, 2018, 251: 86-92.
[49] Abdi H, Williams L J. Principal component analysis[J]. Interdisciplinary Reviews: Computational Statistics, 2010, 2(4): 433-459.
[51] Zhan H L, Xi J F, Zhao K, et al. A spectral-mathematical strategy for the identification of edible and swill-cooked dirty oils using terahertz spectroscopy[J]. Food Control, 2016, 67: 114-118.
[52] Zhang H, Li Z, Chen T, et al. Discrimination of traditional herbal medicines based on terahertz spectroscopy[J]. Optik, 2017, 138: 95-102.
[53] Zou Y, Li J, Cui Y Y, et al. Terahertz spectroscopic diagnosis of myelin deficit brain in mice and rhesus monkey with chemometric techniques[J]. Scientific Reports, 2017, 7(1): 5176.
[54] Lian F Y, Xu D G, Fu M X, et al. Identification of transgenic ingredients in maize using terahertz spectra[J]. IEEE Transactions on Terahertz Science and Technology, 2017, 7(4): 378-384.
[55] Liang J, Guo Q J, Chang T Y, et al. Reliable origin identification of Scutellaria baicalensis based on terahertz time-domain spectroscopy and pattern recognition[J]. Optik, 2018, 174: 7-14.
[56] Kistenev Y, Borisov A, Titarenko M, et al. Diagnosis of oral lichen planus from analysis of saliva samples using terahertz time-domain spectroscopy and chemometrics[J]. Journal of Biomedical Optics, 2018, 23(4): 1-8.
[57] Luo H, Zhu J P, Xu W N, et al. Identification of soybean varieties by terahertz spectroscopy and integrated learning method[J]. Optik, 2019, 184: 177-184.
[58] Zhang P, Zhong S C, Zhang J X, et al. Application of terahertz spectroscopy and imaging in the diagnosis of prostate cancer[J]. Current Optics and Photonics, 2020, 4(1): 31-43.
[60] Zhang Z, Ding H W, Yan X, et al. Sensitive detection of cancer cell apoptosis based on the non-bianisotropic metamaterials biosensors in terahertz frequency[J]. Optical Materials Express, 2018, 8(3): 659-667.
[61] Peng Y, Shi C J, Xu M Q, et al. Qualitative and quantitative identification of components in mixture by terahertz spectroscopy[J]. IEEE Transactions on Terahertz Science and Technology, 2018, 8(6): 696-701.
[62] Du C M, Zhang X, Zhang Z Y. Quantitative analysis of ternary isomer mixtures of saccharide by terahertz time domain spectroscopy combined with chemometrics[J]. Vibrational Spectroscopy, 2019, 100: 64-70.
[63] Zhang X, Lu S H, Liao Y, et al. Simultaneous determination of amino acid mixtures in cereal by using terahertz time domain spectroscopy and chemometrics[J]. Chemometrics and Intelligent Laboratory Systems, 2017, 164: 8-15.
[64] Türker-Kaya S, Huck C W. A review of mid-infrared and near-infrared imaging: principles, concepts and applications in plant tissue analysis[J]. Molecules, 2017, 22(1): 168.
[65] Liu H S, Zhang Z W, Zhang X, et al. Dimensionality reduction for identification of hepatic tumor samples based on terahertz time-domain spectroscopy[J]. IEEE Transactions on Terahertz Science and Technology, 2018, 8(3): 271-277.
[66] Liu W, Zhao P G, Wu C S, et al. Rapid determination of aflatoxin B1 concentration in soybean oil using terahertz spectroscopy with chemometric methods[J]. Food Chemistry, 2019, 293: 213-219.
[67] Huang P, Cao Y, Chen J, et al. Analysis and inspection techniques for mouse liver injury based on terahertz spectroscopy[J]. Optics Express, 2019, 27(18): 26014-26026.
[68] Huang P, Huang Z, Lu X, et al. Study on glycoprotein terahertz time-domain spectroscopy based on composite multiscale entropy feature extraction method[J]. Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy, 2020, 229: 117948.
Article Outline
朱亦鸣, 施辰君, 吴旭, 彭滟. 生物医学检测中太赫兹光谱技术的算法研究[J]. 光学学报, 2021, 41(1): 0130001. Yiming Zhu, Chenjun Shi, Xu Wu, Yan Peng. Terahertz Spectroscopy Algorithms for Biomedical Detection[J]. Acta Optica Sinica, 2021, 41(1): 0130001.