一种基于增强卷积神经网络的病理图像诊断算法  下载: 1243次
下载: 1243次
1 引言
病理切片在疾病诊断中发挥着巨大作用,准确的病理切片诊断能有效协助医生针对疾病制定最佳治疗方案[1],因此病理切片诊断是医学图像分析领域中的一个重要课题。传统人工诊断方法需要医生在显微镜下对细胞的形态等特征进行分析,不仅需要投入大量时间,也容易受经验等主观因素的限制影响诊断结果[2]。为了帮助医生缓解在病理切片诊断上的压力和实现更客观的诊断,人们开始研究使用计算机来协助医生对细胞进行诊断,并设计了计算机辅助诊断(Computer Aided Diagnosis,CAD)系统[3]。CAD系统能发现人眼无法获取的细胞组织内在信息,自动对病理切片中组织是否病变进行诊断,解决了人工病理切片图像诊断中存在的耗时大、主观性强及准确率低等问题。病变与健康组织通常表现为细胞形态上的差异,所以细胞的纹理、大小等形态特征是CAD系统做出准确诊断的重要依据[4-5]。常用的CAD方法主要有稀疏编码和深度神经网络。
稀疏编码是一种有效的特征提取方法,其基本思想是利用一组“超完备”基向量实现对输入样本的稀疏表示。Guo等[6]在稀疏性约束的条件下将细胞的概率分布表征为专家乘积模型,并将多个模型的响应值拼接成用于分类的特征。Vu等[7]提出了可区分的定向特征字典学习算法,针对每一类样本中细胞的形态特征,学习专属于此类样本的稀疏字典,并用稀疏字典对测试样本进行表示。文献[ 8]构建了一个同步的稀疏模型,将训练图像的颜色通道进行稀疏线性组合,通过优化病理图像的稀疏表示来获取用于病理诊断的特征。
深度神经网络能自动学习病理图像中细胞的形态等抽象特征,从而有效识别细胞是否发生病变。卷积神经网络(Convolutional Neural Network,CNN)是视觉信息处理中应用最广泛的一种深度网络结构,是图像分类、图像检索[9]及图像超分辨率研究[10]等领域的重要手段。CNN能通过卷积操作提取病理图像的空间与颜色通道间的非线性关系。Yonekura等[11]用CNN分析脑胶质瘤病理切片图像特征,以实现对肿瘤等级的判断。文献[ 12]利用有标签的成对结肠切片图像训练CNN的特征学习能力,使用训练好的CNN对无标签的图像进行标注,并通过逐渐加入新标注的图像对网络参数进行调整,提升网络对细胞核的识别能力。文献[ 1]中使用GoogleNet[13]学习乳腺癌病理切片图像中的组织抽象特征,并加入数据增强与迁移学习来避免过拟合。褚晶辉等[14]也将CNN与迁移学习进行结合对乳腺癌病理图像进行诊断,获得很好的诊断性能。此外,郑群花等[2]首先采用AlexNet[15]网络对乳腺癌病理切片图像块进行分类,并对多个图像块的分类结果进行多数投票。
上述稀疏特征提取算法主要针对每一类样本提取其中的细胞形态特征,从而实现病理图像的诊断。然而每一类细胞的形态变化多样,而且一些病变细胞与健康细胞的形态非常相似[16],导致算法的诊断准确率较低。此外,基于CNN的病理切片图像诊断算法大多利用单一的网络提取病理切片图像中细胞的特征,而特征提取依赖于网络结构和网络中的卷积核,所以网络结构和卷积核数量的限制导致网络对细胞特征学习具有局限性。不同于以上算法,本文采用补偿的思想,提出一种基于增强卷积网络(BCNN)的病理图像诊断模型。训练一个卷积网络作为基本分类器(BC),对图像中组织发生病变的概率进行估计,以真实标签与概率的差值为目标训练一个增强网络对概率进行补偿。本文在不同类型的病理切片图像上验证了所提模型的性能,该模型对宾夕法尼亚州立大学动物诊断实验室(ADL)提供的数据集中肾、肺、脾三个器官的图像级诊断准确率均在90%以上;对淋巴结癌症转移检测(Camelyon16)数据集的图像块级诊断准确率为93.3%。
2 基于增强卷积网络的病理图像诊断模型
2.1 图像预处理
为了获取病理切片中细胞的局部特征信息,将病理切片图像截取为图像块作为卷积网络的训练样本。在训练图像上以40 pixel为步长,从上到下、从左到右滑动,截取尺寸为80 pixel×80 pixel的图像块用于训练。因为对病理切片染色、扫描及病变区域标注等操作的成本较高,所以可用于训练的病理切片图像的数量有限,而卷积网络需要大量的样本来训练才能准确地提取细胞特征,否则容易出现过拟合现象。为了有效提取细胞特征,本文对训练样本进行扩充,采用随机旋转90°/180°/270°、左下平移截取后再放大、左右及上下镜像4种扩充方式,将样本集扩充为原来的5倍。经过上述方式扩充的图像块与基本图像块相比,只有细胞的空间布局发生改变,其几何形态与颜色等属性保持不变。为了减小因染色造成的色度不同及扫描时光照的变化等因素引起的病理切片图像差异对诊断结果的影响,统一将训练样本归一化为零均值、单位方差。
2.2 增强卷积网络模型
卷积神经网络中的卷积核能够学习病理切片图像中细胞局部区域的非线性关系,并提取人工无法得到的抽象特征,从而实现对病理切片图像的精确识别。因此本文训练一个卷积神经网络作为基本分类器,用以估计细胞发生病变的概率。病理切片图像中的每一类细胞的纹理与大小等变化多样,但是一些患病与健康细胞的形态却非常接近,以上问题增加了深度网络准确识别细胞组织病变与否的难度,使得诊断准确率较低,难以满足临床诊断的高标准要求。常用的解决方法是多次将诊断错误的样本加入训练集后再次训练分类器,增大诊断错误的样本在训练集中所占比重,使得分类器主要针对这些样本进行特征学习。但是多次使用诊断错误样本训练网络容易产生过拟合现象。为此,采用补偿的方法,在基本分类器的基础上,训练一个增强卷积网络对基本分类器的输出值进行修正。所提模型的训练过程如
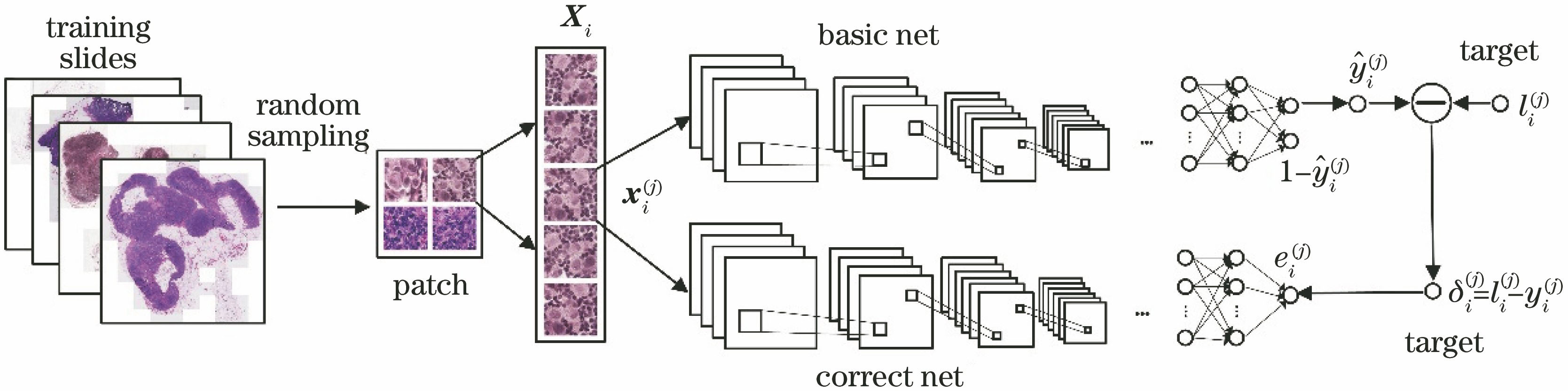
图 1. 增强卷积网络模型的训练过程
Fig. 1. Training process of boosting convolutional neural network model
2.2.1 基本分类器模型
AlexNet在自然图像识别中应用广泛,能够以较少的参数量实现优异的识别性能。病理切片图像虽然与自然图像有明显差异,但是图像中细胞是否发生病变主要反映于细胞质与细胞核的形态变化,而这些形态变化体现在形状、纹理及颜色等特征上。经过自然图像预训练的AlexNet能够学习到自然图像的基本视觉属性,所以网络中的卷积核有助于学习病理图像的细胞形态特征。本文以在ImageNet上预训练的AlexNet的卷积层作为基本分类器的特征提取单元,且在卷积层后设置节点数分别为2048、1000、500、2的4个全连接层,输出层采用Softmax函数输出两个值,一个为组织患病的概率,另一个为组织健康的概率,两概率之和为1。在训练过程中,患病样本的标签为1,健康样本的标签为0。
训练数据集由从病理图像中滑动截取的基本图像块与扩充图像块组成。因扩充图像块的属性与基本图像块完全相同,故与基本图像块有相同的标签。一个理想的分类器对图像样本输出值不会因图像中细胞的空间位置变化而有所不同,所以基本分类器对扩充图像块与基本图像块的输出值应具有一致性。为了衡量这种一致性,在基本分类器的损失函数中加入一项
则
设训练样本集为
式中:
2.2.2 增强卷积网络模型
虽然AlexNet能够对病理图像进行较准确的诊断,但仍存在分类错误的情况,说明AlexNet对图像样本中细胞的特征学习能力具有局限性。为了进一步减少分类错误情况的发生,训练一个增强网络对基本分类器的输出值进行补偿。卷积网络主要依靠卷积核学习病理切片图像中的细胞组织特征,若采用与基本分类器相同的网络作为增强网络,其结构与卷积核数量并未发生变化,可能无法提升性能。因此,为了提高网络的特征学习能力,选择预训练的VGG16[17]的变形网络作为增强网络,实现对基本分类器输出值的修正。
保持VGG16网络的卷积层不变,在卷积层后设置4个全连接层,且前3个全连接层的节点数与基本分类器的前3个全连接层节点数相同。不同于基本分类器,增强网络采用随机纠正线性单元(Leaky-ReLU)激活函数,最后一个全连接层的节点数为1。因为增强网络的训练目标是样本标签与基本分类器输出值之差,所以增强网络的输出层采用Tanh函数输出一个范围为-1~1的修正值。
增强网络的训练样本集同样为
式中:
增强网络训练完成后,以基本分类器与增强网络对图像块的输出值之和作为图像块患病的最终概率值。子集
2.3 病理图像分类
每一个测试图像块经过训练好的基本分类器与增强网络模型的计算后,可分别得到其发生病变的概率值
式中:
为了从图像级判断整张图像是否发生病变,在图像块级的等错误率对应的阈值下,将来自同一张测试图像的图像块进行分类。分类后计算在每张图像上患病图像块所占比例,并以该比例为依据,计算图像级诊断的等错误率,然后以1与等错误率的差值作为图像级的诊断准确率。
3 实验结果
为了验证所提模型的性能,本研究分别在两种不同类型的病理切片图像上进行了测试。由宾夕法尼亚州立大学提供的ADL数据集为哺乳动物的肾、肺、脾三个器官的病理切片图像,淋巴结癌症转移检测数据集Camelyon16为乳腺癌前哨淋巴结病理切片图像。
3.1 ADL数据集实验结果
ADL数据集中包括由肾、肺、脾三种器官的图像构成的三个子数据集,实验时分别在每个子数据集中随机取患病和健康两类图像各40张用于训练[7],110张用于测试。因为数据集中的每张图像尺寸均为1024 pixel×1360 pixel,故在每张图像中共产生782张尺寸为80 pixel×80 pixel的图像块。由于在患病图像中患病细胞占主体,因此在患病图像中截取的图像块都属于患病类。同样地,健康图像中获得的图像块都属于健康类。完成取块后,对两类图像块进行扩充后作为训练样本。
在训练过程中,所提模型中基本分类器的学习率设为5×10-4,训练1.2×104次;增强卷积网络的学习率设为1×10-4,训练4×104次。
实验过程中,分别对3个子数据集训练其相应的增强卷积网络模型,并计算图像块级与图像级的诊断准确率。本实验比较了基本分类器与所提模型对3个子数据集的图像块级与图像级的诊断准确率,对比结果如
表 1. 肾、肺与脾的图像块级与图级诊断准确率
Table 1. Patch-level and image-level diagnosis accuracies of kidney, lung and spleen%
| |||||||||||||||||||||||||||||||||||
当患病与健康两类图像的诊断错误数量之和最小时,实验中计算了所提模型对每个器官患病与健康图像各自的诊断准确率,并与针对ADL数据集实现诊断任务的PoE(Product of Experts)算法[6]和DFDL(Discriminative Feature-Oriented Dictionary Learning)算法[7]的准确率进行比较,不同算法对3个器官的诊断结果如
表 2. 肾、肺与脾图像的诊断结果
Table 2. Diagnosis results of kidney, lung and spleen images%
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
由
3.2 Camelyon16数据集实验结果
为了进一步验证所提算法的性能,将本模型用于图像更复杂的Camelyon16数据集,实现对数据集的图像块级诊断。Camelyon16数据集中的患病切片图像中既包含患病组织,也包含健康组织,且两类组织区域间无明显界限,因此对患病与健康组织的交界处实现准确诊断相对困难。此外,Camelyon16数据集中的病理切片图像由两个机构提供,图像颜色有较大差异,且最大放大倍率下的图像尺寸很大。Camelyon16数据集包含270张训练图像和129张测试图像,其中训练图像中有160张健康图像,110张患病图像;测试图像中有80张健康图像,49张患病图像。该数据集中的每张图像切片在放大40倍下有接近2×1010个像素点,其中将近80%为白色底片背景。
为了减少计算量,先在6级放大倍率下提取切片中的有效组织区域,然后把区域坐标映射到最高放大倍率下进行取块。本研究采取的操作如下:1)对切片的灰度级图像采取Otsu方法[18]得到一个阈值;2)在阈值下将灰度级图像转化为二值图。得到有效组织区域后,将组织区域坐标经过26倍扩大映射到最高放大倍率图像中,并在区域中滑动截取256 pixel×256 pixel的图像块,然后对图像块做分类处理。病变区域面积大于20%的图像块被判为患病样本,其余为健康样本。分类完成后,分别在患病与健康样本中随机采样相同数量的样本,并对样本进行扩充作为训练样本集,扩充方式为随机旋转90°/180°/270°,左下平移截取并放大,左右及上下镜像。
因为网络主要依据图像块中细胞的形状、纹理等特性来实现细胞诊断,图像的缩放不影响上述特性,所以为了减少网络训练时间,将图像块统一缩放为80 pixel×80 pixel的图像块并作归一化处理。基本分类器训练2.6×105次,增强网络训练1.3×105次,其余参数设置与ADL数据集实验相同。每张测试图像被截取为256 pixel×256 pixel的图像块,且所有图像块被缩放为80 pixel×80 pixel后输入到训练好的基本分类器与增强网络中。实验分别计算了所提模型与基本分类器的图像块级诊断准确率,基本分类器的图像块级诊断准确率为91%,所提模型的图像块级诊断准确率为93.3%,比基本分类器提高了2% 以上。实验说明采用异构网络进行特征联合学习,能够补偿单一网络的特征学习能力,提取更准确的细胞抽象特征,使得经过修正后的概率值更接近样本真实标签。
此外,将所提模型与针对Camelyon16数据集实现图像块级诊断的IMSLN(Identifying Metastases in Sentinel Lymph Nodes)[19]与IMBC(Identifying Metastatic Breast Cancer)[20]的图像块级诊断准确率进行了比较,结果如
表 3. Camelyon16数据集中图像的块级诊断准确率
Table 3. Patch-level diagnosis accuracies of images in Camelyon16 dataset%
|
4 结论
为了提高病理切片图像的诊断准确率,提出一种基于增强卷积网络的病理图像诊断模型。本模型训练一个基本分类器对图像块中的组织进行特征学习,并估计组织患病的概率值;以概率值为基础,计算图像块真实标签与概率值的差值,并将此差值作为目标训练一个增强网络,使其输出对概率值的补偿值,以实现对概率值的修正。实验证明增强网络可以克服基本分类器的特征学习局限,实现与基本分类器的特征学习互补。本文在ADL数据集与Camelyon16数据集上验证了本模型的性能。结果表明,所提模型与其他代表性算法相比,能实现更加准确的病理图像诊断。
[1] 何雪英, 韩忠义, 魏本征. 基于深度学习的乳腺癌病理图像自动分类[J]. 计算机工程与应用, 2018, 54(12): 121-125.
[2] 郑群花, 段慧芳, 沈尧, 等. 基于卷积神经网络和迁移学习的乳腺癌病理图像分类[J]. 计算机应用与软件, 2018, 35(7): 237-242.
[4] GianniniV, RosatiS, CastagneriC, et al. Radiomics for pretreatment prediction of pathological response to neoadjuvant therapy using magnetic resonance imaging: Influence of feature selection[C]∥2018 IEEE 15th International Symposium on Biomedical Imaging (ISBI 2018), April 4-7, 2018, Washington, DC, USA. New York: IEEE, 2018: 285- 288.
[5] Alzubaidi AK, Sideseq FB, FaeqA, et al. Computer aided diagnosis in digital pathology application: Review and perspective approach in lung cancer classification[C]∥2017 Annual Conference on New Trends in Information & Communications Technology Applications (NTICT), March 7-9 ,2017, Baghdad, Iraq. New York: IEEE, 2017: 219- 224.
[6] 郭琳琳, 李岳楠. 基于专家乘积系统的组织病理图像分类算法[J]. 激光与光电子学进展, 2018, 55(2): 021008.
[8] SrinivasU, MousaviH, JeonC, et al. SHIRC: A simultaneous sparsity model for histopathological image representation and classification[C]∥2013 IEEE 10th International Symposium on Biomedical Imaging, April 7-11, 2013, San Francisco, CA, USA. New York: IEEE, 2013: 1118- 1121.
[9] 李振东, 钟勇, 陈蔓, 等. 基于深度特征的快速人脸图像检索方法[J]. 光学学报, 2018, 38(10): 1010004.
[10] 李素梅, 雷国庆, 范如. 基于双通道卷积神经网络的深度图超分辨率研究[J]. 光学学报, 2018, 38(10): 1010002.
[11] YonekuraA, KawanakaH, Peasath V B S, et al. Improving the generalization of disease stage classification with deep CNN for glioma histopathological images[C]∥2017 IEEE International Conference on Bioinformatics and Biomedicine (BIBM), November 13-16, 2017, Kansas City, MO, USA. New York: IEEE, 2017: 1222- 1226.
[12] ShaoW, SunL, Zhang DQ. Deep active learning for nucleus classification in pathology images[C]∥2018 IEEE 15th International Symposium on Biomedical Imaging (ISBI 2018), April 4-7, 2018, Washington, DC, USA. New York: IEEE, 2018: 199- 202.
[13] SzegedyC, LiuW, JiaY, et al. Going deeper with convolutions[C]∥2015 IEEE Conference on Computer Vision and Pattern Recognition(CVPR), June 7-12, 2015, Boston, MA, USA. New York: IEEE, 2015: 1- 9.
[14] 褚晶辉, 吴泽蕤, 吕卫, 等. 基于迁移学习和深度卷积神经网络的乳腺肿瘤诊断系统[J]. 激光与光电子学进展, 2018, 55(8): 081001.
[16] Lin HJ, ChenH, DouQ, et al. ScanNet: a fast and dense scanning framework for metastastic breast cancer detection from whole-slide image[C]∥2018 IEEE Winter Conference on Applications of Computer Vision (WACV), March 12-15, 2018, Lake Tahoe, NV, USA. New York: IEEE, 2018: 539- 546.
Article Outline
孟婷, 刘宇航, 张凯昱. 一种基于增强卷积神经网络的病理图像诊断算法[J]. 激光与光电子学进展, 2019, 56(8): 081001. Ting Meng, Yuhang Liu, Kaiyu Zhang. Algorithm for Pathological Image Diagnosis Based on Boosting Convolutional Neural Network[J]. Laser & Optoelectronics Progress, 2019, 56(8): 081001.







